Uma técnica simples para obter DNA genômico a partir de esfregaços nasais adequado para análise de genotipagem em ovinos
DOI:
https://doi.org/10.31285/AGRO.28.1452Palavras-chave:
chip do SNP, esfregaço nasal, extração de DNA, ovinos, análise de concordânciaResumo
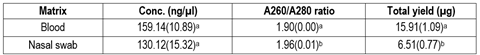
O isolamento do DNA genômico com alta qualidade e quantidade é essencial para estudos moleculares. É crucial selecionar uma técnica não invasiva e simples para garantir a recolha eficiente de ADN, especialmente ao nível das fazendas. O objectivo deste estudo foi determinar se os esfregaços nasais são uma matriz biológica apropriada para obter ADN genómico de boa qualidade adequado para genotipagem de SNP. Neste estudo, duas matrizes biológicas (sangue e esfregaço nasal) foram avaliadas e comparadas para o isolamento de DNA genômico obtido de 15 ovelhas fêmeas Texel. A determinação da qualidade e quantidade do DNA foi realizada por espectrofotometria e eletroforese em gel. As taxas de concordância genotípica foram utilizadas para comparação. Os resultados mostraram que a média de concentração mais alta foi obtida nas amostras de sangue (159,14 ng/µl), enquanto nas amostras de esfregaço nasal a média de concentração foi menor (130,12 ng/µl), mas a diferença não foi significativa. Em relação à pureza, o DNA obtido em esfregaços nasais apresentou maior relação A260/A280 (1,96), enquanto o obtido em amostras de sangue foi de 1,90. O rendimento total de DNA obtido em amostras de sangue (15,91 µg) foi significativamente superior ao obtido em esfregaços nasais (6,51). As taxas de concordância de sangue e esfregaço nasal foram elevadas (média = 0,984). Concluindo, nossos resultados indicam que esfregaços nasais podem produzir DNA de boa qualidade; no entanto, o protocolo de extração de DNA deve ser otimizado.
Downloads
Referências
Aguilar I. SeekParentf90 [Internet]. Georgia: University of Georgia; 2017 [cited 2024 May 03]. Available from: http://nce.ads.uga.edu/wiki/doku.php?id=readme.seekparentf90
Chacon-Cortes D, Griffiths L. Methods for extracting genomic DNA from whole blood samples: current perspectives. Journal of Biorepository Science for Applied Medicine. 2014;2:1-9.Doi: 10.2147/BSAM.S46573. DOI: https://doi.org/10.2147/BSAM.S46573
Chiong KT, Damaj MB, Padilla CS, Avila CA, Pant SR, Mandadi KK, Ramos NR, Carvalho DV, Mirkov TE. Reproducible genomic DNA preparation from diverse crop species for molecular genetic applications. Plant Methods. 2017;13:106. Doi: 10.1186/s13007-017-0255-6. DOI: https://doi.org/10.1186/s13007-017-0255-6
Fan B, Du Z, Gorbach D, Rothschild M. Development and application of high-density SNP arrays in genomic studies of domestic animals. Anim Biosci. 2010;23(7):833-47. Doi:10.5713/ajas.2010.r.03. DOI: https://doi.org/10.5713/ajas.2010.r.03
Foley C, O'Farrelly C, Meade KG. Technical note: Comparative analyses of the quality and yield of genomic DNA from invasive and noninvasive, automated and manual extraction methods. J Dairy Sci. 2011;94(6):3159-65. Doi: 10.3168/jds.2010-3987. DOI: https://doi.org/10.3168/jds.2010-3987
Hawken RJ, Cavanagh JA, Meadows JR, Khatkar MS, Husaini Y, Zenger KR, McClintock S, McClintock AE, Raadsma HW. Technical note: Whole-genome amplification of DNA extracted from cattle semen samples. J Dairy Sci. 2006;89(6):2217-21. Doi: 10.3168/jds.S0022-0302(06)72292-5. DOI: https://doi.org/10.3168/jds.S0022-0302(06)72292-5
Lee RA, Herigon JC, Benedetti A, Pollock NR, Denkinger CM. Performance of Saliva, Oropharyngeal Swabs, and Nasal Swabs for SARS-CoV-2 Molecular Detection: a Systematic Review and Meta-analysis. J Clin Microbiol. 2021;59(5):e02881-20. Doi: 10.1128/JCM.02881-20. DOI: https://doi.org/10.1128/JCM.02881-20
Li G, Gelernter J, Kranzler HR, Zhao H. M(3): an improved SNP calling algorithm for Illumina BeadArray data. Bioinformatics. 2012;28(3):358-65. Doi: 10.1093/bioinformatics/btr673. DOI: https://doi.org/10.1093/bioinformatics/btr673
Masuda Y, Legarra A, Aguilar I, Misztal I. 331 Efficient quality control methods for genomic and pedigree data used in routine genomic evaluation. J Anim Sci. 2019;97(Suppl 3):50-1. Doi: 10.1093/jas/skz258.101. DOI: https://doi.org/10.1093/jas/skz258.101
Medrano JF, Aasen E, Sharrow L. DNA extraction from nucleated red blood cells. Biotechniques. 1990;8(1):43.
Murphy MA, Shariflou MR, Moran C. High quality genomic DNA extraction from large milk samples. J Dairy Res. 2002;69(4):645-9. Doi: 10.1017/s0022029902005848. DOI: https://doi.org/10.1017/S0022029902005848
Neary MT, Neary JM, Lund GK, Garry FB, Holt TN, Mohun TJ, Breckenridge RA. Technical note: A comparison of DNA collection methods in cattle and yaks. J Anim Sci. 2014;92(9):3811-5. Doi: 10.2527/jas.2013-7445. DOI: https://doi.org/10.2527/jas.2013-7445
Núñez L, Rodríguez MI, Giménez G, Martínez R. Evaluación y comparación de dos protocolos de extracción de ADN a partir de tres tipos de muestras de ovinos. Compend Cienc Vet. 2022;11(2):18-23. Doi: 10.18004/compend.cienc.vet.2021.11.02.18. DOI: https://doi.org/10.18004/compend.cienc.vet.2021.11.02.18
Psifidi A, Dovas CI, Banos G. A comparison of six methods for genomic DNA extraction suitable for PCR-based genotyping applications using ovine milk samples. Mol Cell Probes. 2010;24(2):93-8. Doi: 10.1016/j.mcp.2009.11.001. DOI: https://doi.org/10.1016/j.mcp.2009.11.001
Psifidi A, Dovas CI, Bramis G, Lazou T, Russel CL, Arsenos G, Banos G. Comparison of eleven methods for genomic DNA extraction suitable for large-scale whole-genome genotyping and long-term DNA banking using blood samples. PLoS One. 2015;10(1):e0115960. Doi: 10.1371/journal.pone.0115960. DOI: https://doi.org/10.1371/journal.pone.0115960
R Core Team, 2021. R: A language and environment for statistical computing [WWW Document]. R Found Stat Comput. URL https://www.R-project.org (accessed 4.29.22).
República Oriental del Uruguay, Poder Legislativo.Procedimientos para la utilizacion de animales en actividades de experimentacion, docencia e investigacion cientifica. Ley N° 18.611 [Internet]. 2009 [cited 2024 May 03]. Available from: https://www.impo.com.uy/bases/leyes/18611-2009
Sambrook J, Russell DW. The condensed protocols from molecular cloning: a laboratory manual. Cold Spring Harbor: Cold Spring Harbor Laboratory Press; 2006. 800p. DOI: https://doi.org/10.1101/pdb.prot3919
Vignal A, Milan D, SanCristobal M, Eggen A. A review on SNP and other types of molecular markers and their use in animal genetics. Genet Sel Evol. 2002;34(3):275-305. Doi: 10.1186/1297-9686-34-3-275. DOI: https://doi.org/10.1051/gse:2002009
Yang W, Kang X, Yang Q, Lin Y, Fang M. Review on the development of genotyping methods for assessing farm animal diversity. J Anim Sci Biotechnol. 2013;4(1):2. Doi: 10.1186/2049-1891-4-2. DOI: https://doi.org/10.1186/2049-1891-4-2
Downloads
Publicado
Como Citar
Edição
Seção
Licença
Copyright (c) 2024 Agrociencia Uruguay

Este trabalho está licenciado sob uma licença Creative Commons Attribution 4.0 International License.
| Métricas do artigo | |
|---|---|
| Vistas abstratas | |
| Visualizações da cozinha | |
| Visualizações de PDF | |
| Visualizações em HTML | |
| Outras visualizações | |